已解决
clusterProfiler包学习
来自网友在路上 184884提问 提问时间:2023-11-19 13:52:13阅读次数: 84
最佳答案 问答题库848位专家为你答疑解惑
📖 Introduction | Biomedical Knowledge Mining using GOSemSim and clusterProfiler (yulab-smu.top)
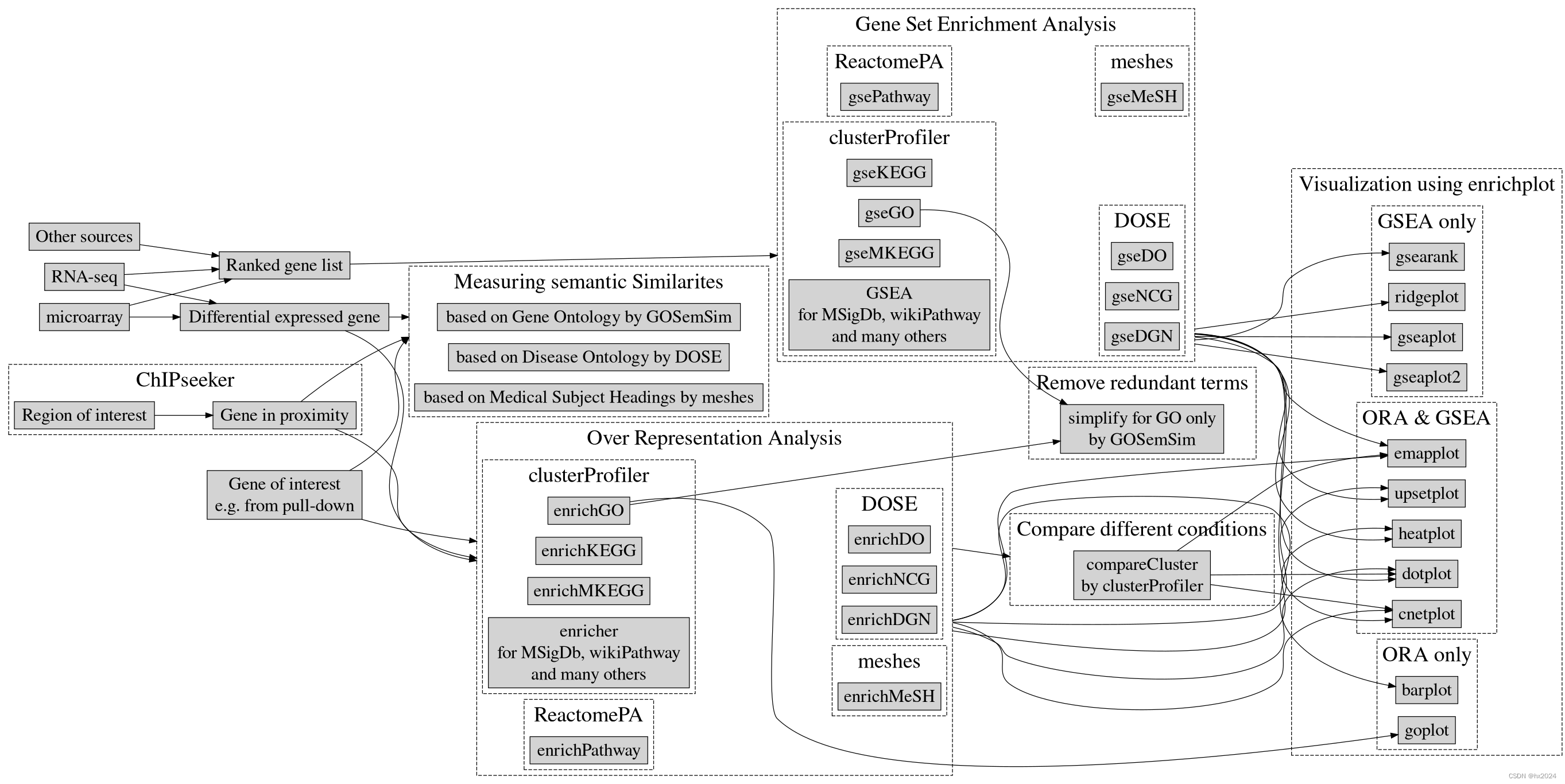
部分使用
#GO classificationlibrary(clusterProfiler)
data(geneList, package="DOSE")
gene <- names(geneList)[abs(geneList) > 2]# Entrez gene ID
head(gene)
ggo <- groupGO(gene = gene,OrgDb = org.Hs.eg.db,ont = "CC",level = 3,readable = TRUE)head(ggo)##############################################################
##KEGG##
library(clusterProfiler)
search_kegg_organism('ece', by='kegg_code')
ecoli <- search_kegg_organism('Escherichia coli', by='scientific_name')
dim(ecoli)data(geneList, package="DOSE")
gene <- names(geneList)[abs(geneList) > 2]kk <- enrichKEGG(gene = gene,organism = 'hsa',pvalueCutoff = 0.05)
head(kk)【精选】RNA 11. SCI 文章中基因表达富集之 GSEA_gsea数据库-CSDN博客
查看全文
99%的人还看了
相似问题
- 基因预测软件prodigal的使用
- 【佳学基因检测】Node.js中如何读取并调用内容
- 零代码复现-TCGA联合GEO免疫基因结合代谢基因生信套路(一)
- 泡泡玛特首度跨界超跑品牌兰博基尼汽车,以潮流基因探索时空边界
- LeetCode:2003. 每棵子树内缺失的最小基因值(C++)
- 易基因: Nature Biotech:番茄细菌性青枯病的噬菌体联合治疗|国人佳作
- 重测序基因组:Pi核酸多样性计算
- GEO生信数据挖掘(三)芯片探针ID与基因名映射处理
- 8+单基因+细胞凋亡+WGCNA+单细胞+实验验证
- DeepMind 利用无监督学习开发 AlphaMissense,预测 7100 万种基因突变
猜你感兴趣
版权申明
本文"clusterProfiler包学习":http://eshow365.cn/6-39316-0.html 内容来自互联网,请自行判断内容的正确性。如有侵权请联系我们,立即删除!
- 上一篇: 【IPC】消息队列
- 下一篇: 宏--offsetof使用